本文转载自:PYNQ开源社区微信公众号
感兴趣者可与 pynq_china@xilinx.com 联系,共同合作拓展项目。
冬天快要到了,细菌们到了卷土重来的季节,那么为了针对细菌的抗生素耐药性,我们该怎么快速地发现对应的药物呢?来自思克莱德大学(University of Strathclyde)的Ryan Greer在OpenHW2020中,利用了PYNQ上的支持向量机,通过使用分类算法,从来自抗生素生产菌的高光谱图像中快速识别细菌菌株的特性。
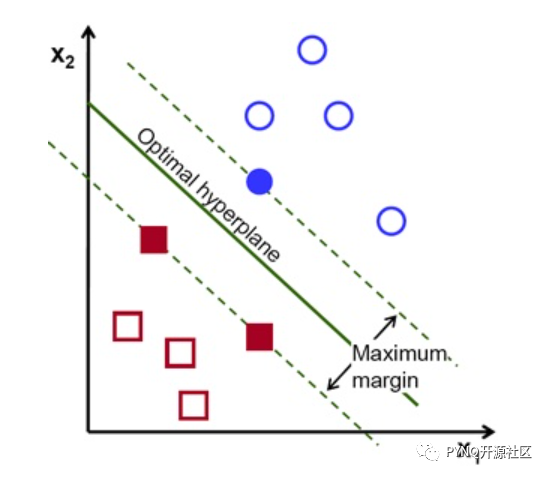
本篇文章介绍一个使用FPGA实现的SVM训练和预测模型。支持向量机(Support Vector Machine)是一种二分类模型,从本质来说,是一种用一条线(方程)分类两种事物的模型。
如下图所示,对于给定的数据集,SVM的任务就是找到一条分界线或分界面(Optimal hyperplane)使得它到两边的距离(margin)都最大。算法详情可以参考 https://en.wikipedia.org/wiki/Support_vector_machine. 本项目使用细菌的高光谱影像作为数据集,对于训练和预测的部分分别进行了硬件设计,最终实现在pynq-z2上。项目地址为https://github.com/RyanMan1/PYNQ-SVM-OpenHW-2020,使用的文件为DEMO文件夹下的jupyter notebook。
1.数据处理
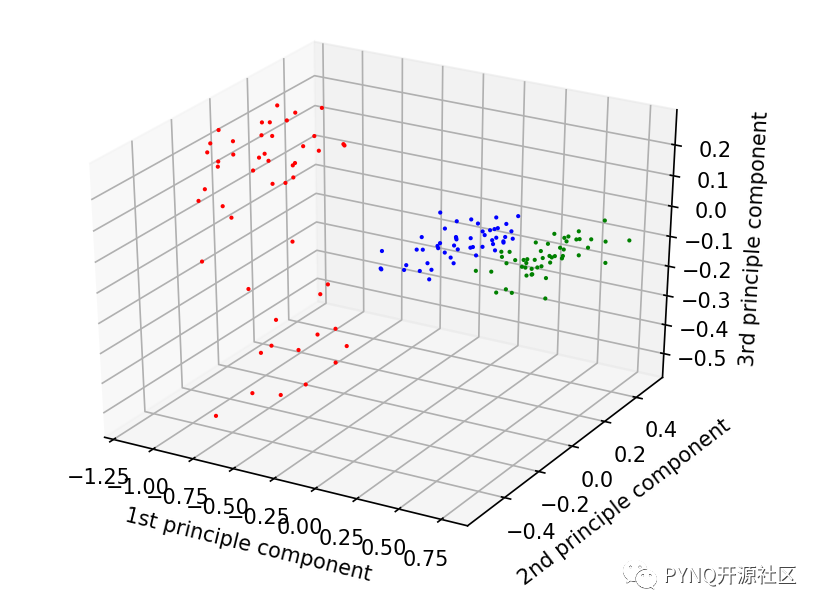
项目中所使用的数据集为细菌的高光谱影像,所谓高光谱影像是指使用特殊的传感器在一个很长的波段上捕获物体的光谱信息。项目中使用的高光谱影像中的每一个像素包含了256个不同波长的信息,整张高光谱图片可以表示成一个数据方块,如下图。每个数据被提前处理成矩阵的形式,矩阵的行代表每一个像素,列代表不同的波长信息。

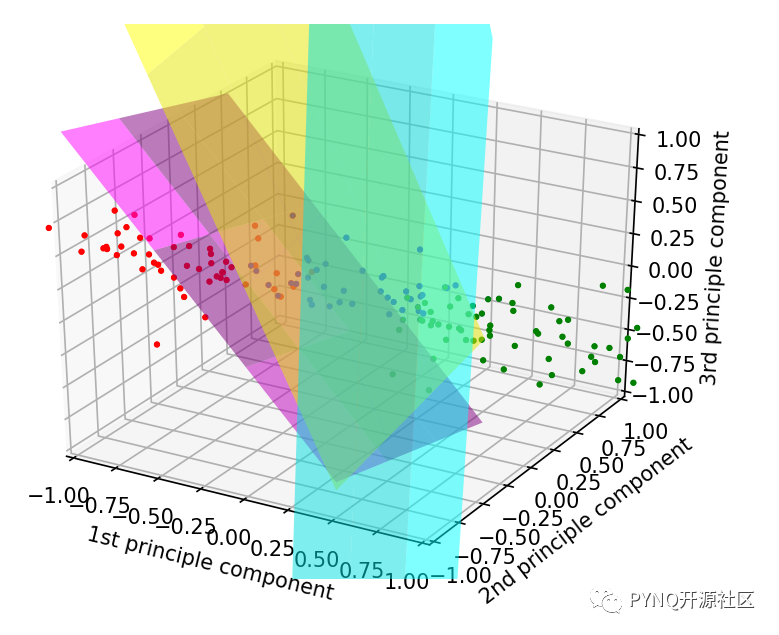
由于原数据是一个256维的向量空间,为了可视化数据,作者使用主成分分析(PCA)将数据降到3个维度,如图.可以看出数据集大概有三个不同的种类。
2.模型训练
首先加载bitstream

开始训练,模型同时实现了线性和非线性的SVM核,其中对于线性的SVM,可以在数据集上做出划分平面来可视化。下图中的三个平面分别代表三个种类
两两之间的分界面。

3.模型预测
首先加载部署模型的bitstream
然后对模型进行测试
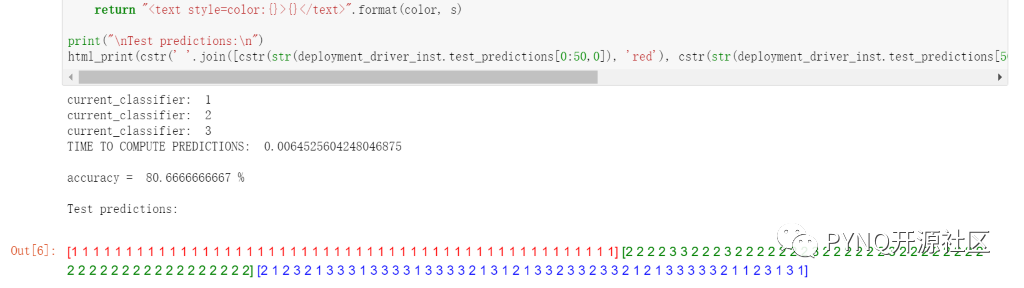
将测试集和预测结果可视化
4.性能分析
作者分别在不同的平台(pynq-z2, zcu104,MATLAB)上进行测试,包括模型的训练,线性和非线性SVM核的预测。得到如下的测试结果
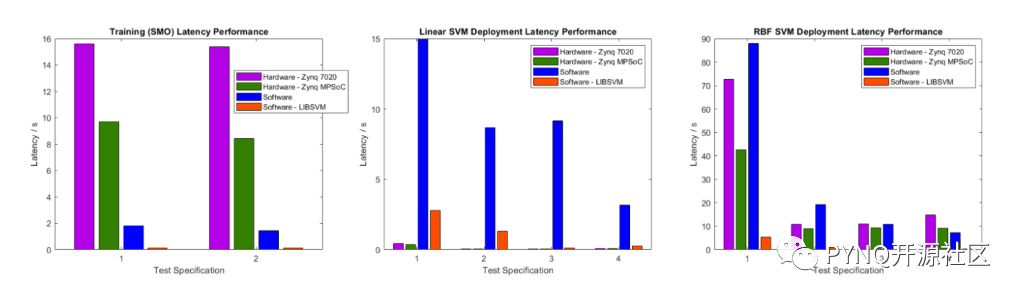
三张图从左到右分别是模型训练,线性SVM和非线性SVM的延时。通过比较发现,对于模型的训练,FPGA的表现不佳是因为数据的读取占用了大部分的时间,而对于SVM预测的计算,FPGA的表现要高于CPU。对于线性的SVM核,甚至跟工业级别的I7处理器以及优秀的MATLAB算法相比,FPGA依然实现了显著的加速。
详细内容可点击github链接查看:https://github.com/RyanMan1/PYNQ-SVM-OpenHW-2020/blob/master/DEMO/Ryan%2...